MAP Themen Flashcards
1
Q
Zellhülle der gram+ Bakterien
A
- Peptidoglykan (Murein)
- Cytoplasmamembran
- Proteine
2
Q
Zellhülle - Peptidoglykan/Murein
A
- in g+ und g-
- Grundstruktur der bakteriellen Zellhülle
- stabilisierende und schützened Exoskelettstruktur
3
Q
Peptidoglykan/Murein - Funktionen
A
- Stabilität
- ZW, verantwortlich dafür, dass die Zelle dem hohen intrazellulären Druck standhält
- Formgebung
- Feste ZW kompensiert für Flexibilität der Phospholipidmembran
- Bestimmt Form der Zelle
- Unbegrenztes Wachstum
- Ständige Vergrößerung und Teilung
- Stoffwechselaktives Kompartiment
4
Q
Peptidoglykan/Murein - Aufbau, BEstandteile
A
- 2 Bestandteile
- Glykan-Rückgrat (x2)
- N-Acetylglucosamin G, N-Acetylmuraminsäure
- Beta-1,4-glycosidische Verbindung à Angriffsstelle für Lysozym, Liquidität, Turgordruck, Zelle platzt
- Peptidbrücke
- Verbindet Glykanstränge
- Muraminsäure
- Glykan-Rückgrat (x2)
- Bibasische Aminosäure (DAP/Lys) ermöglicht tail to tail Verknüpfung der Peptidreste
- Der Glykanteil variiert in Bakterien nur geringfügig (O oder N-Acetylkation)
- Der peptidteil kann sich insbesondere zwischen
-
Quervernetzung
- Enzymatische Transpeptidierung
- Energiebereitstellung von Abspatung Alaninrest
- Knüpfung D-Ala-DAP-Bindung
5
Q
Zellhülle - Proteine
A
- Braunsche Lipoproteine
- Verankerung des Mureins mit der Außenmembran über das Braunsche Lipoprotein
- Abundantes Protein
- Häufig
- Kovalent mit Peptidteil verbunden
6
Q
Zellhülle der gram- Bakterien
A
- Äußere Membran
- LPS
- Peptidoglykan/Murein
- Cytoplasmamembran
- Proteine
7
Q
Zellhülle gram- Bakterien - Äußere Membran (LPS)
A
- Äußere Membran
- Assymetrisch (innen Phospholipide, außen LPS)
- Diffusionsbarriere für große Moleküle (u.a. viele Antibiotika)
- Permeabel für kleinere hydrophile Moleküle (bis ca. 600 Da)
-
Lipopolysaccharid (LPS)
- A Teil
- Essentiell
- Überlebenswichtig
- Kernpolysaccharid
- Seitenketten
- Variabel
- O-Spezifische Polysaccharide
- Funktion
- Schutz
- Strukturelle Integrität
- Erhöht negative Ladung der Membran
- Adhäsion an Oberflächen
- Sensititvität gegenüber Bakteriophagen
- Relevant in Pathogenität von gram- bakterieen (Antigenwirkung, Endotoxin)
- A Teil
8
Q
Wachstumskurve einer statischen Kultur
A
- Nur ein Nährmedium
- Es wird kein neues Medium hinzugefügt bei Verbrauch
- Messung optische Dichte einer Bakterienssuspension
- Wachstumsrate proportional zur optischen Dichte
- Lag phase: Neusynthese von Transportproteinen und Enzymen
- Log phase: hauptsächlich Ribosome
- Post-__exponentielle Phase: Flagellen, Chemotaxis
- Entwicklung der Zellzahl in der exponentiellen Phase:

9
Q
Binäre Zellteilung
A
- Teilen in der Mitte
- Bildung Septum in der Mitte
- Einstülpung Zellwand
10
Q
E. coli Zellteilung
A
- Längenwachstum
- Repliziert Chromosom
- Trennung deer Chromosomen, Nukleoide
- Voraussetzung für nächsten Schritt
- Z-Ring-Bildung
- FtsZ: Protein, filamentous temperature sensitive
- Signal für den nächsten Schritt
- Divisombildung
- Proteinkomplex zuständig für Peptidoglykansynthese
- Wird an FtsZ-Ring rekrutiert
- Einschnürung Septum
- Teilung

11
Q
FtsZ
A
- Protein für Zelllteilung
- Filamentous temperature sensitive
- Bei 30°C funktional
- WT bei 42° normal
- Ftsz Mutante wird lang, kein Septumbildung
- Tubulin-homolog
12
Q
MinCDE System
A
- Erkennung der Zellmitte
- Mutanten der Gene können nicht Septum in der Zellmitte bilden
- Können Mitte nicht lokalisieren
- Minizellen beinhalten keine DNA
Vermehrung nicht möglich
13
Q
MinCDE Komponenten
A
- MinC
- Inhibitor der Z-Ring Bildung
- MinD
- Bildet Membrananker für MinC
- ATPase Aktivität
- MinCD bildet in vivo einen heterodimeren Komplex
- MinE
- Verdrängt MinCD von der Membran
- Möglicherweise durch Auflösung des Heterodimers
14
Q
MinCDE -Oszillation
A
- Mechanismus der Zellmitte-Lokalisierung und Septumbildung in der Zellmitte
- System oszilliert zwischen den Polen
- wandern von einen Zellpol zur nächsten
- MinC-GFP markiert
- MinE-GFP markiert
- Modellaufstellung
- Lokalisierung FtsZ, MinCD und MinE
- MinCD wandert zu Polen, gefolgt von MinE, welches MinCD verdrängt
- über Zeit gesehen in der Mitte der Zelle Konzentration von MinCD am geringsten
- so kann in der Mitte FtsZ ausbilden
15
Q
Bedeutung der protonenmotorischen Kraft
A
- Elektronentransport
- Respiration
- Photosynthese
- Transport
- Flagellenbewegung
- ATP Herstellugn
- Biosynthesen
- Transport
16
Q
Zuckertransporter in E.coli
A
- Maltose-ABC Transporter
- ABC Transporter
- Lactose-System
- PEP-PTS
- Phosphoenolpyruvat-Phosphotransferasesystem
- Dient der Hexose-Aufnahme, vorwiegend Glucose
17
Q
Maltose-ABC Transporter
A
- Maltase/Maltodextrin-Transport von E. Coli
18
Q
Maltose-ABC-Transporter - Struktur
A
- Rot: Periplasma
- Bindeprotein
- Substratgebundener Zustand bindet an Transmembrandomäne
- Blau und Gelb: innere Membran
- Transmembrandomäne
- Lila, Grün: Cytosol
- ATP-Bindedomäne

19
Q
Maltose-ABC_Transporter Funktionsweise
A
- Maltosebindeprotein und Maltose im Periplasma
- Bindung Maltose und Maltosebindeprotein à Konformationsänderung zu geschlossenen Zustan
- Bindung geschlossene Transmembrandpmän ATP bindet MalK
- Konformationänderung der ATP-Domäne à Konformationsänderung der Transmembrandomäne (Kanalproteine)
- Durch ATP-Hydrolyse Originalzustand der Proteine

20
Q
Lactose-System
A
- Sekundärer Transportsystem
- PMF-abhängige Symbporter
21
Q
Lactose-System Struktur
A
- Blau: Lactose
- Transporter 1 Protein
- 12 Transmembrandomänen

22
Q
Lactose-System Funktionswweise
A
- Protonengradient (Atmungskette)
- Lactosepermease (LacY) bindet Lactose und Proton
- Lässt Lactose und Proton ins Cytoplasma
- H+ kannin ETK wieder in Periplasma eingeschleust werden

23
Q
PEP-PTS
A
- Gruppentranslokation –
- Phosphoenolpyruvat – Phosphotransferase System
24
Q
PEP-PTS Komponenten
A
- Enzym I à unspezifisch
- Enzym II (A,B,C) à Spezifisch
- Histidinprotein à unspezifisch
25
PEP-PTS Funktionsweise
* Phosphatrest von PEP auf EI übertragen
* Übertragung auf HPr
* Übertragung aus E II
* Phosphorylierung des Substrats aus Periplasma in EIIC

26
PTS
* Phosphorylierungs-Reaktion ist nicht wie bei Phosphokinasen ATP und Mg2+, sondern PEP-abhängig
* Enzym E I = durchgreführte Reaktion ist pleiotrop = steht generell allen PTS-Zucker Transporten zur Verfügung
* Von E II katalysierte Reaktion ist eine spezifische Reakiton = jeder PTS-Zucker hat ein eigenes E II
* Mutationen in HPr oder E I = unspezifische Auswirkungen, d.h. kein PTS-Zucker kann mehr verstoffwechselt weren
* Mutationen in E II = spezifisch, d.h. es ist immer nur ein PTS-Zucker-Stoffwechselweg betroffen
27
Energiegehalt in PTS-Systemen
* Die Phsophorylierung besitzen vom PEP bis E II B den gleichen Energiegehalt
* Befinden sich nahezu im GGW
* Erst bei Phosphorylierung des Substrates (PTS-Zucker) findet starker Energieabfaöö statt
* Nur in Gegenwart von PTS-Zuckern wird das Reaktionsgleichgewich nach rechts gezogen
28
* Beispiele für PTS Zucker
* Glucose
* Fructose
* Trehalose
* Mannitol
* GluNAC
* Mannose
29
* Nicht-PTS-Zucker
* Lactose
* Maltose
* Arabinose
* Galactose
* Ribose
* Xylose
30
Bacteriorhodopsin
* Nicht in Bakterien, sondern Halophilen Bakterien
* Integrales Membranprotein
* Protonenpumpendes Chromoprotein
* Sekundärstrukturelemente
* 7 Helikale Bereiche, die die Membran durchspannen
* In 7. Transmembran Helix ist an Lysinrest _Retinal_ gebunden
* Retinal
* All-trans-Retinan
* Cofaktor
* Salzkonzentrationen an Sättigungsgrenze
* Aerobier
* Nutzen Bacteriorhodopsin unter Sauerstoffmangel
* Große Mengen an Bacteriorhodopsin in Membran, purpur

31
Bacteriorhodopin - Lichtreaktion
* Nach Richtreaktion all-trans-Retinal à 13-cis-Retinal
* Protonierte Schiffsche Base ihre Position in Protein verändert von Innenseite zur Außenseite wander
* Proton wird zunächst aus saure AS übertragen und auf Außenseite freigesetzt
* Retinalcofaktor geht wieder in Grundzustand über
* Saure AS wird mit H versorgt
* Geht in all-trans Konfiguration über
32
Anaerobe Fütterungsketten
Respiration ohne Sauerstoff
* Links:
* ausreichend Sulfat als Elektronenakzeptor
* Sulfatreduzenten spielen entscheidende Rolle
* Vollständige Mineralisation des Kohlenstoffs bis zum CO2
* Rechts
* Monomere zu Gärungsprodukten umgesetzt
* Syntrophe (auf Partner angewiesen) Gärer, die primäre Gärungsprodukte weiter zu Acetat verstoffwechseln
* Homoacetogenese C1-Verbindungen, H2 zu Acetaat

33
^Gärung
* In Abwesenheit von externen Elektronenakzeptoren, daher keine E-Transportkette
* Ausscheidung noch relativ energiereicher reduzierter Endprodukte
* Organische Säuren und/oder Ethanol
* Daneben Freisetzung von CO2 und H2
* Unvollständiger Abbau von Zuckern unter anaeroben Bedingungen
* Vermeidung von Reduktionsäquivalenten

34
Alkoholische Gärung
* Abbau von Glucose unter anaeroben Bedingungnen zu Ethanol
* Energiegewinnung 2 ATP aus Glykolyse
* Regeneration des Cofaktors NAD+

35
Sulfatreduktion - Typen
* Unvollständige Oxidierer
* Vollständige Oxidierer
* Autotrophe
36
Sulfatreduktion - Unvollständige Oxidierer
* Oxidieren organische Säuren über Pyruvat zu Acetyl-CoA und scheiden Acetat aus
37
Sulfatreduktion - Vollständige Oxidierer
* Oxidieren Fettsäuren, KH oder Aromaten über Acetyl-CoA bis zum CO2
* Bei Biomineralisation von organischen Verbindungen besonders wichtif
* In marineen Umgebungen
* Sulfatkonzentration höher im Meer
* Akkumullierung Biomasse in Gewässer
* Biomasse in obere Zone kann oxisch umgesetzt werden --\> aerobe Atmung Mineralisierung
* In tiefen Shcichten
* Gärung
* Acetat als Hauptprodukt
* Kann auch oxidiert werden
* Verstoffwechseln Gärungsendprodukte
38
Sulfatreduktion - Autotrophe
* Nutzen H2 als E-Quelle und fixieren CO2 über den Acetyl-CoA-Weg oder den reduktiven Trikarbonzyklus (nur bei Anaerobiern)
39
Schritte der Sulfatreduktion
* Sulfat gelangt in Zelle über Symport mit Protonen (1)
* Energieverbrauch
* Aktivierung Sulfat mithilde ATP-Sulfurylase (2)
* Mit ATP zu APS
* Pyrophosphatase in zwei mol. Anorg. Phosphat
* GGW in Richtung APS
* APS durch APS-Reduktase 2e- Schritt zu Sulfit (3)
* AMP wird wieder freigestzt
* AMP mit ATP zu 2 ADP
* 2 ADP mit 2 Pi über ATPSynthase zu 22 ATP phosphorylierten (8)
* 6 Proteonen verbrauch
* 2 E-reiche Bindungen wurden verbraucht, 2 E Bindungen geknüpft à keine E-Konservierung! Protoenenpotential
* Aufbau Protoenenpotential Möglichkeit
* H2 als e-Donator und Protoenen Quelle, DH spaltet 4 Moleküle (5)
* 8 Protonen
* 6 für ATP-Synthase
* 2 für Symport
* En
* Energetisches Nullsystem
* Pyrophosphatasen bauen Protonenpotential auf

40
Anoxygene Phototrophe Bakterien
* Unterschiedliche Elektronendonatoren
* Z.B. Schwefelwasserstoff
* Keine O2 Bildung
* PS I ODER PS II ohne Wasserspaltung
* Ernährungsweisen
* unterschiedlich
41
Beispiele Anoxygene Phototrophe Bakterien
* Purpurbakterien (Schwefel und Nicht-Schwefel)
* Grüne Schwefelbakterien
* Grüne Nicht-Schwefel Bakterien
* Heliobakteriien
42
Winogradsky-Säulen - Anreicherungsverfahren für anoxygenen phototrophen Bakterien
* **_K_**ulturen in Glassäulen
* Winogradsky-Säulen
* von oben belichtet
* ansonsten abgedeckt
* Nicht-Schwefelpurpurbakterien
* Befüllung
* Proteinlösung -\> Erde -\> Sand
* Begießen Gewässerprobe
* ganzeoben auf Deckel Lichtfilter
* bei 800-900 nm: Bakterien mit Bchl a
* bei 900-1100 nm: Bakterien mit Bchl b
* Schwefelpurpurbakterien und Grüne Schwefelbakterien
* Befüllung
* Faulschlamm, Gips, Erde
* Umgebung die Sulfatatmung begünstig
* Freiisetzung H2S
* Schwefelpurpur
* mag nicht zu hohe Konzentrationen H2S, Schicht daher etwas oberhalb
* Grüne Schwefelbakterien
* üblicherweise ausgeprägt resistent höherer Konzentrationne
* Wachstum direckt überhalnn

43
Purpurbakterien
* Schwefel, nicht-Schwefel
* RC durch ringförmig angeordnete Proteinkomplexe umgeben
* diese PK enthalten BChl a, Bezeichnung LH

44
Grüne Schwefelbakterien
* obligat photolithoautotroph
* H2S als e-Donator für PS
* reduktiven Zitronensäurezyklus als CO2 Fixierung
* große Chlorosomen
* bis zu 250 000 BChl Moleküle
* keine direkte Interaktion mit RC
* BChl a bindenendes Protein (FannerMatthews-Olson) FMO Protein zwischengeschaltet
* Basalschicht weggelassen
* Spektrale Eigenschaften der Pigmente in jeweiligen Ketten (?)
* erlauben Energietransfer von den Pigmenten in AS bis RC
* BChl c (742nm) ,d,e in Chlorosomen in Rotbereich kurzwelligere Anregungswellenlängen als BChl a in Basalplatte
* BChl a in Basalplatte (792 nm) kurzwelliger als BChl a in FMO Proteinen
* BChl a in FMO (805 nm) kurzwelliger als im RC (865 nm)
Energiegefälle, sodass thermodynamisch möglich vorgeschaltete Pigmente ihre E an nachgeschaltete Pigmente weiterzugeben
45
Grüne Nicht-Schwefel Bakterien
* photolithoautotroph oder chemoorganoheterotroph
* CO2 Fixierung: 3 Hydroxypropionatzyklus
* Wasserstoff als e-Donator oder org. Verb.
* kleine Chlorosomen
* bis zu 50 000 BChl Moleküle
46
Heliobakterien
* keine CO2 Fixierungsweise
* Ernährung
* Lebensweise Photoorganoheterotroph (oder Chemo)
* obligate anaerobier
* nacktes Homodimeres RC
* keinerlei Antennensysteme nachgewiesen
47
Reaktionszenrten
* Komplexe von integralen Membranproteinen
* Stattfindungsort der Ladungstrennung
* Nach Lichtabsorption wird Elektron auf ein negatives Redoxppotential gebracht
* nur 1 PS in jeweiligen Organismen
48
PS I
* Homodimere
* Fe-S-Zentren als Elektronencarrier
* 4F-4S-Zentrum
* Cystein-Schwefel
* 1 der 4 Eisenionen Valenzwechsel durchlaufen (+3 à +2 bzw. +2 à +3)
49
PS II
* Heterodimere (dunkel und hellmagenta)
* Chinon (Q) als Elektronencarrier
* Fettlösliche Elektronencarrier
* 2 Carbonylgruppen
* Einzeln nacheinander reduziert werden können
50
Merke
* Das Redox-Potential des reduzierten Fe-S-Akzeptors von Typ I-RC ist deutlich negativer als das des Chinonakzeptors von Typ II-RC
51
Antennen
* Pigment
* Versch. Varianten von Antennensystemen,, da RZ alleine nicht ausreichend
* Unterschiedliche AS in unterschiedlichen Organismen sorgen für effizientes Lichtsammeln
* Chlorosomen
* Lichtsammel(antennen) Komplexe der Grünen Bakterien
* Lipidmonoschicht Kompartimente
* riesege Anzahl v bChl c, d und e eingeschlossen
* Größe kann variieren
52
Räuber unter gramnegativen Bakterien
Beispiel: *Bdellovibrio bacteriovorus*
53
*Bdellovibrio bacteriovorus*
* Halbmondförmig
* Bakterien fressender Blutsauger
* Annährung an stäbchenförmige Zelle
* Schwimmt frei herum bis mit Beute zusammenstößt
* Bohrt in Wirtszelle hinein
* Verlust des Falgellums
* Entry in periplasmatischen Raum
* Vergrößerung des Räubers
* Abrundung Wirtszelle à Bdelloplast
* Räuber teilt sich in mehrere kleine Zellen
* Lyse der Beutezellwand, Platzen, Freisetzen der geteilten Räuberzellen
54
Bacteriocine
* für Baktereien toxisch Peptide
* Ribosomal synthetisierte Peptide, die Prokaryoten ausscheiden und die andere Prokaryoteen durch unterschiedlche Mechanismen abtöten
* Eine Klasse bilden die Lantibiotika, Peptide, die die ungewöhnliche Aminosäure Lanthionin enthalten
* Lanthioninringe entstehen in Petpiden in denen Cysteinreste mit mod. Serinresten oder Threoninresten, die mehrere AS-Reste entfernt sind, Schwefelbrücke ausbilden
* Keine Redoxlabile Disulfidbrücke sonder Verknüpfung

55
Bacteriocine Beispiel Nisin
* Lactococcus Lactis
* Nisin A wirkt gegen gram+ Bakterien
* Gram+ Krankheitserreger in Rohmilchprodukten werden abgetötet von Nisin A à macht diese Produkte für uns menschen erträglich
* _Wirkmechanismus_
* N-Terminus bindet an Lipid 2, Vorstufe bei ZW Synthese, an Außenseite
* Lipophiler Carrier + Disaccharid + Pentapeptid à Lipid II
* Verhinderung weitere ZW Synthese
* Keine Neuen Zuckerbindungen
* Nisin A an Lipid 2 an Außenseite wird festgehalten
* Begünstigt Insertion der C-terminalen Domäne in die Membran
* C-terminale Domönene Oligomerisieren, bilden Pore in Membran à Potentiale brechen zusammen
56
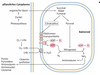
Symbiontische Bakterien in Wurzelknöllchen
* Stickstofffixierung
* Pflanzen scheiden Flavonoide aus (Startsignal)
* Luteolin
* Genistein
* Verbindungen mit 2 aromatischen Ringen
* Bakterien sensen die Flavonoide
* Produktion Nodulationsfaktoren
* Enthält 3 Kopien N-Acetyl-Glucosamin
* Eines Sulfatiert
* Pflanze reagiert mit Ausbildung Infektionsschlauch
* Bakterien können durch Infektionsschlauch ins Innere der Wurzell eindringen
* In Pflanzenwurzel werden B einzeln in gruppen von Pflanzenmembran umgeben
* Bakteroide ändern Form
* Werden zu stickstoffixierenden Sklaven ausgebildet
* Wechselwirkung der Pflanzen und Bakteroide
57
Pflanzenpathogene Bakterien
* *Agrobacterium tumefaciens*
* *Pectobacterium carotovorum*
* *Erwinia amylovora*
* *Pseudomonas syringae*
58
*Agrobacterium tumefaciens*
* Wurzelhalstumoren

59
*Pectobacterium carotovorum*
* Abbau der Zellwandkomponente Pektin
* Verlust der Stab. Der pflanzl. ZW
* Kartoffel, Karotten etc.
60
*Erwinia amylovora*
* Feuerbrand
* Abfallen und Bräunung der Blätter
* Über Blüte in Pflanze mithilfe bestäubende Insekten
* Wandert durch Leitbündel
* Systemische Vermehrung innerhalb ganze Pflanze
* Produziert Exopolysaccharide à verstopft Leitbündel
* StofftrANSport unterwindet
* Exopolysaccharide ziehen Insekten an, Zyklus
61
*Pseudomonas syringae*
* epiphylle bakteriuen
* Infizieren Blätter
* Über trichome oder geöffnete Stomata
* In intrazellulären Raum
* Typ III-Sekretionssysteme
* Massenhafte Vermehrung
* Effektorproteine inserieren
* Nekrosen
* Ice Nucleation
* Kristallationskeim
* An bohnen


