VL 3: Stoffwechsel I Flashcards
(47 cards)
1
Q
Transportsysteme
A
- äußere Membran der gram- Bakterien als Barriere
- mit Porinen
2
Q
Porine - LamB E.coli Maltoporin
A
- zuständig für Aufnahme Maltose
- Beta-Faltblattstruktur charakteristisch für Porine
- Trimer
- LamB
- Bakteriophage lambda
- Rezeptor
- Diffusionsaufnahme

3
Q
Diffusion und aktiver Transport
A
Diffusion
- Passiver Transport
- Polare Verbindungen (Fettsäuren)
- Kleine polare Substanzen (Wasser, Ethanol, Glycerol, Harnstoff
- Gase
- Cytoplasmamembran ist impermebael für größere polare Substanzen (Glucose und Ionen
Aktiver Transprortprozess
- Diffusion abhängig von Konzentration außerhalb
- Aktiver Transport besser bis zu bestimmter Konzentration à Sättigung
- Spezifisch
- Benötigen Energie
- Spez aktiven häufig stark reguliert
- Genexpression
- Aktivität

4
Q
Aktive Transportsysteme
A
- Primäre Transportsysteme
- nutzen chemische Energie
- ATP Hydrolyse
- Sekundäre Transportsysteme
- nutzen Ionengradien
- Meist 12 Transmembrandomänen
- verbrauchen weniger Energie
- geringere Affinität(Spzifiät)
- hohe Transportrate

5
Q
Beispiele Primäre Transportsystem
A
- ATP-Synthase
- ETK
- Ione-ATPasen
- ABC-Transporter
- ECF
- Decarbxylasen
- PTS

6
Q
ABC-Transporter
A
- ATP-binding-cassette
- Transport v. Zuckern, AS, anorganischen Substraten (z.B. Sulfat, Phosphat) und Spurenelemente
- Hohe Substratspezifität
- Periplasmatisches Bindeprotein
7
Q
ECF
A
- Energy-coupling factor
- ABD Transportter ohen extrazelluläres Bindeprotein
- Besitzen Transmembranuntereinheit zur Substratbindung
- Ni2+/Co2+ oder wasslösliche Vitmaine (Biotin, Riboflavin, Thiamin
8
Q
Decarboxylasen
A
- Koppeln Biotin-abhängige Decarboxylierung von Carbonsäuren wie Oxalat mit dem Export von Na+ IOnen
9
Q
PTS
A
- Gruppentranslokation
- Phosphoenolpyruvat-Phosphotransferasesystem
- Phosphorylgruppe von PEP (verfügt über hohes Gruppenübertragungspotential) wird über mehrere Proteinkinasen auf das Substrat (Hexosen und Zuckeralkohole (Mannitol übertragen)
10
Q
Beispiele Sekundäre Transportsysteme
A
- TRAP-Transporter
- Uniport
- Symport
- Antiport

11
Q
TRAP-Transporter
A
- Tripartite ATP-independent periplasmic-Transporter
- Weit verbreitet in Prokaryoten, jedoch nicht in eukaryotischen Zellen
- C4-Dicarbonsäuren, Verbindungen für Osmoregulation (Ectoin, Taurin)
12
Q
Maltose-ABC-Transporter
A
Maltase/Maltodetrin-Transport von e.coli
13
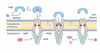
Q
Maltose-ABC-Transporter - Struktur
A
- Rot: Periplasma
- Bindeprotein
- Substratgebudn. Zustand bindet an Transmembrandomäne
- Blau und gelb: innere Membran
- Transmembrandomäne
- Cytosol: lila grün
- ATP bindemonäne

14
Q
Maltose-ABC-Transporter Funktionsweise
A
- Maltosebindeprotein und Maltose im Periplasma
- Bindung Maltose und Maltosebindeprotein à Konformationsänderung zu geschlossenen Zustand
- Bindung geschlossene Transmembrandomäne, ATP bindet an MalK
- Konformationsänderung der ATP-Domäne à Konformaitonsänderung der Transmembrandomäne (Kanalprotene)
- Durch ATP-Hydrolyse Originalzustand der Proteine
15
Q
Lactose-System
A
- Sekundärer Transportsystem
- PMF-abhängige Symporter
16
Q
Lactose-system Struktur
A
- Blau: Lactose
- Transporter 1 Protein
- 12 Transmembrandomänen

17
Q
Lactose-System Funktionsweise
A
- Protonengradient (Atmungskette)
- Lactosepermase (LacY) bindet Lactose und Proton
- Lässt Lactose und Proton ins Cytoplasma
- H+ kann in ETK wieder in Periplasma eingeschleust werden

18
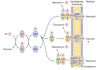
Q
Gruppentranslokation - PEP-Phosphotransferase-System (PTS)
A
- Verschiedene Enzyme
- Unspezifische Komponenen
- Enzym I
- Histitidprotein
- Spezifität
- EII Komponenten (A,B,C)
- EIIC Transmembran
- EIIA und B Fusionen

19
Q
PEP-PTS Funktionsweise
A
- Phosphatrest von PEP auf EI übertragen
- Übertragung auf HPr
- Übertragung auf E II
- Phosphorylierung des Substrats aus Periplasma in EIIC
20
Q
PTS
A
- Phosphorylierungs-Reaktion ist nicht wie bei Phosphokinasen ATP und Mg2+, sondern PEP-abhängig
- Enzym E I = durchgreführte Reaktion ist pleiotrop = steht generell allen PTS-Zucker Transporten zur Verfügung
- Von E II katalysierte Reaktion ist eine spezifische Reakiton = jeder PTS-Zucker hat ein eigenes E II
- Mutationen in HPr oder E I = unspezifische Auswirkungen, d.h. kein PTS-Zucker kann mehr verstoffwechselt weren
- Mutationen in E II = spezifisch, d.h. es ist immer nur ein PTS-Zucker-Stoffwechselweg betroffen
21
Q
Energiegehalt in PTS-Systmen
A
- Die Phsophorylierung besitzen vom PEP bis E II B den gleichen Energiegehalt
- Befinden sich nahezu im GGW
- Erst bei Phosphorylierung des Substrates (PTS-Zucker) findet starker Energieabfaöö statt
- Nur in Gegenwart von PTS-Zuckern wird das Reaktionsgleichgewich nach rechts gezogen

22
Q
Beispiele PTS-Zucker
A
- Glucose
- Fructose
- Trehalose
- Mannitol
- GluNAC
- Mannose
23
Q
Nicht-PTS-Zucker
A
- Lactose
- Maltose
- Arabinose
- Galactose
- Ribose
- Xylose
24
Q
Regulation
A
- Nährmedien mit versch. Zuckern
- Bakterium kann sich aussuchen
- Regulation !
25
Diauxie
* Wachstum auf Mischung von Glucose und Lactose
* Zuerst wachsen Bakterien auf Glucose
* Lagphase nach verbrauch der Glucose
* Anschließendes Wachsen auf Lactose
* Wachstum schneller auf Glucose
* Lactose muss gespaltet werden
* Blaue linie: Exprimieren der beta Galactosidase
* ZP: bei Verbrauch von Glucose -à daher Lagphase

26
Das *lac* Operon Funktion
* 3 Gene unter Kontrolle eines Promotors
* *lacZ, lacY, lacA*
* Protranskribierung der 3 Gene
* Entehung polycistronische mRNA
* Riibosomen binden mRNA
* Tranlation
* Transkription und Translation können gleichzeitig stattfinden
* Proteine
* Beta-glactosidase (LacZ)
* Permease (LacY)
* Transactylase (LacA)

27
Regulation des *lac* Operons
* Repression
* Induktion
28
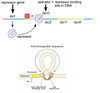
Regulation des *lac* Operons - Repression
* Repressorprotein codiert von Gene *lacI*
* Repressor: Bindeprotein
* Bindet Operator *lacO* (repressor bindestelle in der DNA)
* Tetramer
* Dimer or dimers
* Bindet an zwei Palindromische DNA Sequenzen, relativ weit weg voneinander
* Sekundärstruktur loop
* *Lac* Operon kann nicht translatieren
29
Regulation des lac Operons - Induktion
* Inducer, Molekül bindet an Represso
* Konf änderung des Repressors
* Abfallen des Repressors

30
Lactose Analoga
* Isopropyl-beta-D-thiogalactopyranosid (IPTG)
* Künstlicher Induktor des *lac* Operons
* O-Nitrophenyl-beta-D-galactopyranosid (ONPG)
* Chromogenes Substrat für beta-Galactosidasen
* Spaltung
* Enzymaktivität Galactosidase messbar
* Verwendung für versch. Sachen
* Allolactose
* Transglycosylierung von Lactose durch LacZ
* Umwandlung der Gla-beta-1,4-Glc-Bindung in eine Gla-beta-1,6-Glc-Bindung
* Physiologische Inucer von LacI
31
Katabolitrepression
* Leicht metabolisierbare C-Quellen wie Glucose, hemmen die Verwertung von Nicht-PTS-Zuckern
* Bei Anwesenheit von Glucoose ist der cAMP-Spiegel niedrig (second messenger)
* Transkriptionsfaktor CAP im Komplex mit cAMP aktiviert Promotoren von nicht PTS-Zuckern
32
cAMP
* globaler second-messenger
* high [Glucise] = low [cAMP]
* low [Glucose] = high [cAMP]
* bindet an Transkriptionsfaktor CAP (catabolite aktivator Protein)
CAP bindet an Promotor, agiert als Aktivator
33
Katabolitrepression - Kontrolle des *lac* Operons
Anfang Wachstumskurve
* Nur Glucose vorhanden
* cAMP nicht an CAP gebunden
* kein Promotoraktivierung
* lac Repressor ist an operator gebinden, weil kein Inducer vorhanden

34
Katabolitrepression - Kontrolle des *lac* Operons - erste Phase Wachstum, G und L vorhanden
* cAMP immernoch gering, keine Aktivierung
* lac Repressor wird
* gewisse Transkription findet statt
* Allolactose
* Nicht bedeuteden

35
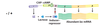
Katabolitrepression - Kontrolle des lac Operons - G verbraucht, L vorhanden
* Bildung cAMP
* Bindung mit CAP
* Transkription nicht verhindert
* Lac Operon
36
Induktorausschluss (Katabolitinhibition)
* Reduktion der Transportkapazität von Nicht-PTS-Zuckern bei Anwesenheit von Glucose
* Dieser soogenannte Induktorausschluss (engl. Inducer Exclusion) verhindert, dass nicht-PTS Systeme indukziert werden, solange genügend Glucose aufgenommen werden kann
* **Adenylatcyclase (Cya)**
* Bildet cAMP
37
Katabolitrepression und Induktorausschluss bei Vorhandensein von Glucose
* Wenn Phosphattransfer zur Glucose stattfindet, wird das GGW zwischen unphosphoryliertem EIIAGlc und phosphoryliertem EIIAGlc(-P) zugunsten unphosphorylierten Form verschoben
* EIIA Glc interagiert direkt mit vielen nicht-PTS-Transportern (z.B. ABC-Transportern oder Protonensymportern) und inhibiert deren Aktivität = **Induktorausschluss**

38
Katabolitrepression und Induktorausschluss bei keiner Glucose
* Wenn Glucose aufgebraucht ist, wird das GGW zwischen unphosphoryliertem EIIAGlc und phosphoryliertem EIIAGlc(-P) zu gunsten der phosphorylierten Form verschoben
* EIIAGlc-P aktiviert die denylatcyclase (Cya)
* cAMP in Komplex mit CAP aktiviert die Expression von nicht-PTS-Zuckertransportern
* Aktivierung von Cya durch EIIAglc-P schaltet Katabolitrepression ab

39
Übersicht über zellulären Stoffwechsel
* Katabolismus
* Substrate
* Umwandlung zu Produkten
* Energieerzeugung
* Umwandlung der Energie zum ATP generieren oder Als protonmotive force
* Anabolismus
* Energieverbauch
* Biosynthese bausteine
40
ATP - Adenosintriphosphat
* Regeneration von ATP
* Substratkettenphosphorylierne
* Oxidativer Abbau organischer Verindungen
* Elektronentransportphosphorylierung
* Atmungskette
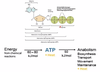
41
Prozesse der Energiegewinnugn

42
Klassifizierung von Organismen nach Energie- und Kohlenstoffquellen
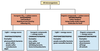
43
Chemoorganotrophes Wachstum - Abbau von Hexosen
asjf
44
Wichtigste Glucose-Abbauwege bei Prokaryoten
* EMP-Weg (Glykolyse)
* KDPG-Weg (Entner-Doudoroff)
* Pentose-Phosphatweg
45
EMP-Weg
1. Phosphorylierung Glucose durch Hexokinase zu G-6-P (=Aktivierung, stoffwechselaktive Form und Ausgangspunkt fü weiterre Abbauwege)
2. Isomerisierung zu F-6-P
3. Phosphorylierung F6P durch Phosphofructokinase zu F16BP
4. Spaltung zu DHAP und GA3P durch Aldolase
5. Dehydrogenierung GAP zu 3-Phosphoglycerat
* wichtigster Schritt
* Abspaltung Hydridanion (H-), Übertragung auf NAD+ und gleichzeitig Entfernung eines H+ = Entstehung von NADH + H+
* Generierung von ATP durch Phosphorolyse und Übertragung auf ADP = reversibel
6. Umwandlung von 3-Phosphoglycerat in 2-Phosphoglycerat
7. Wasserabspaltung durch Enolase von 2-Phosphoglycerat zu PEP
8. Energiereiche Phosphrylgruppe von PEP (Enolester durch Pyruvat-Kinase auf ADP übertragen
Pyruvat ist Vorstufe weiterer Abbau Umwandlung und Syntheseprozesse.
bilanz: 2 Pyruvat, 2 ATP, 2 NADH + H+
46
47
Schlüsselreaktionen der E-gewinngung im EMP-Weg
2. Herkunft von Reduktionsäquivalenten (NADH+H+)
4. beispiel für Substratstufenphospphorylierung



